如何用 QuPath 轻松实现生物医学图像分析?2024 完整指南
QuPath 是一款免费开源的生物医学图像分析软件,专为病理学家、研究人员和科学家设计,可高效处理显微镜图像、组织切片等生物医学影像数据。无论是癌症研究中的细胞计数、组织微阵列分析,还是药物研发中的图像量化,QuPath 都能提供直观且强大的工具支持,帮助用户快速从复杂图像中提取有价值的信息。
🧠 QuPath 核心功能:不止是图像查看器,更是分析利器
QuPath 整合了多种先进算法与可视化工具,让生物医学图像分析变得简单高效。以下是其最值得关注的核心功能:
✅ 全切片图像与显微镜图像处理
支持多种图像格式(如 TIF、VSI、APERIO、Hamamatsu 等),可流畅加载和浏览超高分辨率的病理切片图像,轻松放大至单个细胞级别观察细节。
✅ 智能细胞分割与组织分析
内置自动化细胞检测与分割工具,可精准识别细胞核、细胞质及细胞膜结构,支持brightfield(明场)和荧光图像的批量分析,显著减少手动标注时间。
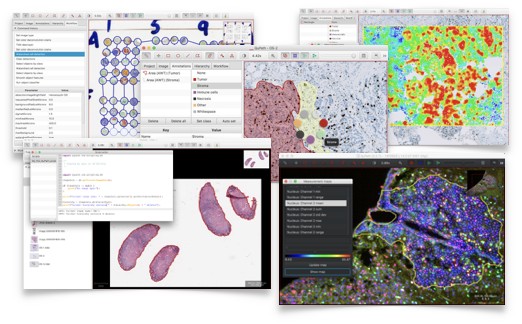
图:QuPath 对病理切片图像的细胞分割与标记效果,不同颜色代表不同类型细胞(QuPath 官方示例图)
✅ 交互式机器学习分类
无需编程基础即可训练自定义分类模型!通过手动标注少量样本,QuPath 可自动学习特征并对图像中的细胞、组织类型进行分类,准确率媲美专业分析软件。
✅ 脚本自动化与批量处理
支持 Python 和 Groovy 脚本编写,可定制化分析流程,实现批量图像的自动化处理、数据导出与报告生成,适合高通量实验数据分析。
✅ 与 ImageJ 无缝集成
可直接调用 ImageJ 的图像处理功能,或导出分析结果至 ImageJ 进一步编辑,兼顾灵活性与专业性。
🔬 3 大典型应用场景:解决你的研究痛点
QuPath 已被全球数万名科研人员用于各类生物医学研究,以下是其最受欢迎的应用方向:
1️⃣ 癌症研究:快速评估肿瘤微环境
- 自动识别肿瘤细胞与免疫细胞,计算肿瘤浸润淋巴细胞(TILs)密度,辅助免疫治疗疗效评估。
- 空间分布分析:量化癌细胞与周围组织的空间关系,揭示肿瘤侵袭模式。
2️⃣ 组织微阵列(TMA)分析
- 自动去阵列化:批量识别 TMA 芯点,提取每个样本的图像数据并统一分析。
- 高通量数据对比:快速比较不同患者样本的病理特征,加速生物标志物发现。
3️⃣ 药物研发与毒理学研究
- 药效评估:量化药物处理后细胞形态变化(如大小、形状、染色强度)。
- 自动化计数与统计:生成标准化数据报告,支持 Excel、CSV 等格式导出,便于后续统计分析。
🚀 新手入门:3 步开启 QuPath 分析之旅
1️⃣ 下载与安装(完全免费!)
访问 QuPath 最新发布页,根据操作系统选择对应版本(Windows/macOS/Linux),解压后即可运行,无需复杂配置。
2️⃣ 快速上手核心功能
- 导入图像:点击菜单栏「File > Open」,选择病理切片或显微镜图像。
- 基础标注:使用工具栏的矩形、多边形工具手动绘制感兴趣区域(ROI)。
- 自动分析:尝试「Analyze > Cell Detection」一键检测细胞,调整参数优化结果。
3️⃣ 学习资源推荐
- 官方文档:QuPath 中文教程(含入门指南与高级技巧)。
- 社区支持:在 Image.sc 论坛 提问,获取开发者与资深用户的实时帮助。
💡 为什么选择 QuPath?5 大优势对比其他工具
| 特性 | QuPath | 传统付费软件(如 HALO、ImageScope) | 基础图像工具(如 ImageJ) |
|---|---|---|---|
| 价格 | 完全免费(开源 GPLv3) | 年费数千至数万美元 | 免费 |
| 生物医学专业性 | ✅ 专为病理图像优化 | ✅ 功能全面但复杂 | ❌ 需插件扩展 |
| 易用性 | ✅ 图形界面+自动化工具 | ❌ 学习曲线陡峭 | ❌ 多步骤手动操作 |
| 批量处理 | ✅ 脚本支持+批量任务 | ✅ 支持但需付费 | ❌ 有限支持 |
| 社区与更新 | ✅ 活跃开发+学术背书 | ✅ 商业支持但更新慢 | ✅ 插件丰富但分散 |
📚 结语:让图像分析不再成为研究瓶颈
无论是初入实验室的研究生,还是资深病理学家,QuPath 都能通过直观的界面和强大的功能,帮助你从生物医学图像中快速挖掘有价值的数据。其开源特性确保算法透明可复现,避免「黑箱分析」带来的结果不确定性。
立即下载 QuPath,开启你的高效生物医学图像分析之旅吧!如有疑问,欢迎加入 QuPath 中文社区 与全球用户交流经验。
提示:如果你的研究使用了 QuPath,请引用其官方论文:Bankhead et al., Scientific Reports (2017),以支持开源项目的持续发展!
 GLM-5智谱 AI 正式发布 GLM-5,旨在应对复杂系统工程和长时域智能体任务。Jinja00
GLM-5智谱 AI 正式发布 GLM-5,旨在应对复杂系统工程和长时域智能体任务。Jinja00 GLM-5-w4a8GLM-5-w4a8基于混合专家架构,专为复杂系统工程与长周期智能体任务设计。支持单/多节点部署,适配Atlas 800T A3,采用w4a8量化技术,结合vLLM推理优化,高效平衡性能与精度,助力智能应用开发Jinja00
GLM-5-w4a8GLM-5-w4a8基于混合专家架构,专为复杂系统工程与长周期智能体任务设计。支持单/多节点部署,适配Atlas 800T A3,采用w4a8量化技术,结合vLLM推理优化,高效平衡性能与精度,助力智能应用开发Jinja00 请把这个活动推给顶尖程序员😎本次活动专为懂行的顶尖程序员量身打造,聚焦AtomGit首发开源模型的实际应用与深度测评,拒绝大众化浅层体验,邀请具备扎实技术功底、开源经验或模型测评能力的顶尖开发者,深度参与模型体验、性能测评,通过发布技术帖子、提交测评报告、上传实践项目成果等形式,挖掘模型核心价值,共建AtomGit开源模型生态,彰显顶尖程序员的技术洞察力与实践能力。00
请把这个活动推给顶尖程序员😎本次活动专为懂行的顶尖程序员量身打造,聚焦AtomGit首发开源模型的实际应用与深度测评,拒绝大众化浅层体验,邀请具备扎实技术功底、开源经验或模型测评能力的顶尖开发者,深度参与模型体验、性能测评,通过发布技术帖子、提交测评报告、上传实践项目成果等形式,挖掘模型核心价值,共建AtomGit开源模型生态,彰显顶尖程序员的技术洞察力与实践能力。00 Kimi-K2.5Kimi K2.5 是一款开源的原生多模态智能体模型,它在 Kimi-K2-Base 的基础上,通过对约 15 万亿混合视觉和文本 tokens 进行持续预训练构建而成。该模型将视觉与语言理解、高级智能体能力、即时模式与思考模式,以及对话式与智能体范式无缝融合。Python00
Kimi-K2.5Kimi K2.5 是一款开源的原生多模态智能体模型,它在 Kimi-K2-Base 的基础上,通过对约 15 万亿混合视觉和文本 tokens 进行持续预训练构建而成。该模型将视觉与语言理解、高级智能体能力、即时模式与思考模式,以及对话式与智能体范式无缝融合。Python00 MiniMax-M2.5MiniMax-M2.5开源模型,经数十万复杂环境强化训练,在代码生成、工具调用、办公自动化等经济价值任务中表现卓越。SWE-Bench Verified得分80.2%,Multi-SWE-Bench达51.3%,BrowseComp获76.3%。推理速度比M2.1快37%,与Claude Opus 4.6相当,每小时仅需0.3-1美元,成本仅为同类模型1/10-1/20,为智能应用开发提供高效经济选择。【此简介由AI生成】Python00
MiniMax-M2.5MiniMax-M2.5开源模型,经数十万复杂环境强化训练,在代码生成、工具调用、办公自动化等经济价值任务中表现卓越。SWE-Bench Verified得分80.2%,Multi-SWE-Bench达51.3%,BrowseComp获76.3%。推理速度比M2.1快37%,与Claude Opus 4.6相当,每小时仅需0.3-1美元,成本仅为同类模型1/10-1/20,为智能应用开发提供高效经济选择。【此简介由AI生成】Python00 Qwen3.5Qwen3.5 昇腾 vLLM 部署教程。Qwen3.5 是 Qwen 系列最新的旗舰多模态模型,采用 MoE(混合专家)架构,在保持强大模型能力的同时显著降低了推理成本。00
Qwen3.5Qwen3.5 昇腾 vLLM 部署教程。Qwen3.5 是 Qwen 系列最新的旗舰多模态模型,采用 MoE(混合专家)架构,在保持强大模型能力的同时显著降低了推理成本。00- RRing-2.5-1TRing-2.5-1T:全球首个基于混合线性注意力架构的开源万亿参数思考模型。Python00